Enkelt-nukleotidpolymorfi (SNP): Definition og rolle i genetik & medicin
Lær hvordan SNP'er påvirker genetik, sygdomsrisiko og skræddersyet medicin — essentiel viden om diagnostik, forskning og behandling.
Enkelt nukleotidpolymorfi (SNP, udtales snip; flertal snips) er en enkel variation i DNA-sekvensen i en population. En SNP er ændringen af ét enkelt nukleotid i genomet — for eksempel kan DNA-sekvenser fra to personer være AAGCCTA og AAGCTTA, hvor de kun adskiller sig ved ét nukleotid. I dette tilfælde siger man, at der findes to alleler. Næsten alle almindelige SNP'er har kun to alleler, og de fleste ligger i ikke-kodende dele af genomet, hvor de ofte ikke påvirker organismens overlevelse og derfor ikke fjernes hurtigt af naturlig udvælgelse.
Forekomst og årsager
SNP'er opstår ved fejl i DNA-replikationen eller som følge af miljøpåvirkninger, og deres tætheden i genomet påvirkes af faktorer som mutationstakt, genetisk rekombination, genetisk drift og selektion. Nogle SNP'er er almindelige i hele verden, andre er hyppige i bestemte befolkningsgrupper. En variant, der er almindelig i en geografisk eller etnisk gruppe, kan være sjælden i en anden — dette har betydning for populationsturisme og medicinsk tolkning.
Funktionel betydning
SNP'er kan være funktionelt neutrale eller have effekt på geners funktion og regulering. Man skelner typisk mellem:
- synonyme SNP'er i kodende regioner, som ikke ændrer aminosyresammensætningen,
- non-synonyme (missense eller nonsense), som ændrer eller stopper proteinets aminosyresekvens,
- SNP'er i regulatoriske regioner, som kan påvirke geners udtryk, og
- SNP'er, der ændrer splitsites eller påvirker ikke-kodende RNA, og dermed kan regulere genfunktion indirekte.
Anvendelser i forskning, sundhed og retssager
SNP'er er centrale i moderne genetik og medicinsk forskning:
- Genome-wide association studies (GWAS) bruger millioner af SNP-markører til at finde varianter, der er associeret med sygdomsrisiko eller andre træk.
- Farmakogenetik undersøger, hvordan SNP'er i fx CYP- eller VKORC1-gener påvirker, hvordan personer metaboliserer lægemidler og dermed dosis eller bivirkningsrisiko.
- SNP-profiler bliver også brugt i populationsgenetik og slægtsforskning for at kortlægge oprindelse og migration.
- I retsvidenskaben anvendes genetiske markører — primært STR'er, men også SNP-paneler i visse situationer — til identifikation og slægtskabsanalyse, især når DNA er svært nedbrudt.
Metoder til påvisning
SNP'er kan påvises ved flere teknologier: målrettet PCR og sekventering, microarray-baserede genotyping-kits og moderne højkapacitets næste-generations-sekventering (NGS). Valg af metode afhænger af formålet — om man vil undersøge nogle få kendte varianter, lave et helt genom eller genotype mange prøver hurtigt og billigt.
Genetisk kobling og tolkningsbegrænsninger
SNP'er optræder ofte i blokke (haplotyper) på grund af linkage disequilibrium; det betyder, at en påvist SNP-association ikke nødvendigvis er årsagen til en effekt, men kan markere en nærliggende, funktionel variant. Mange sygdomme er multifaktorielle, hvor mange SNP'er hver især bidrager med en lille effekt, og miljøfaktorer også spiller en stor rolle.
Etik, privatliv og klinisk relevans
Brug af SNP-data rejser spørgsmål om fortrolighed, genetisk diskrimination og tolkning af risici. En positiv association mellem en SNP og en sygdom betyder ikke nødvendigvis, at en person får sygdommen — ofte er risikoændringen relativt lille. Derfor kræves forsigtig formidling og, når relevant, genetisk rådgivning ved klinisk brug.
Samlet set er SNP'er et grundlæggende værktøj i moderne genetik: de hjælper os med at forstå genetisk variation, sygdomsrisiko, lægemiddelrespons og befolkningshistorie, men kræver omhyggelig metode, tolkning og etisk håndtering.
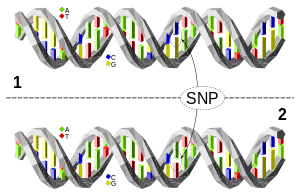
DNA-molekyle 1 adskiller sig fra DNA-molekyle 2 på et enkelt basepar (en C/A-polymorfi)
Spørgsmål og svar
Q: Hvad er en SNP?
A: En enkeltnukleotidpolymorfisme er en DNA-sekvensvariation i en population, der er karakteriseret ved en enkelt nukleotidforskel i genomet.
Q: Hvad står SNP for?
A: SNP står for single nucleotide polymorphism (enkeltnukleotidpolymorfisme).
Q: Hvor mange alleler har almindelige SNP'er?
A: De mest almindelige SNP'er har kun to alleler.
Q: Hvor forekommer SNP'er oftest i DNA'et?
A: SNP'er forekommer oftest i områder af DNA'et, som ikke påvirker organismens overlevelse.
Q: Hvilke faktorer kan påvirke SNP-tætheden?
A: Faktorer som genetisk rekombination og mutationsrate kan også påvirke SNP-tætheden.
Q: Hvad er nogle af de praktiske anvendelser af genetiske variationer mellem individer?
A: Genetiske variationer mellem individer (især i ikke-kodende dele af genomet) udnyttes nogle gange i DNA-fingeraftryk, som bruges i retsmedicinsk videnskab. Disse genetiske variationer forårsager også forskelle i vores modtagelighed over for sygdomme.
Q: Hvad er et eksempel på en genetisk variation, der er forbundet med en højere risiko for en bestemt sygdom?
A: For eksempel er en enkeltbasemutation i APOE-genet (apolipoprotein E) forbundet med en højere risiko for Alzheimers sygdom.
Søge